2023年11月,瞿介明教授课题组在《Genome Medicine》杂志发表题名为“Mobilizable plasmids drive the spread of antimicrobial resistance genes and virulence genes in Klebsiella pneumoniae”——肺炎克雷伯菌诱动转移型质粒促进耐药基因和毒力基因的播散的研究论文。该研究揭示了肺炎克雷伯菌诱动转移型质粒转移的分子机制和影响因素:揭示了诱动转移型质粒可以逃避受体菌CRISPR-Cas系统的攻击,并且相比接合型质粒携带更少的原间隔序列和具有更广的宿主范围,提示了诱动转移型质粒对于促进耐药基因和毒力基因播散中的重要贡献。这项研究是基于瞿介明教授课题组2021年发表在《Genome Medicine》杂志题为“Mobilization of the nonconjugative virulence plasmid from hypervirulent Klebsiella pneumoniae”的研究工作上,所作出的进一步探讨。
瑞金医院呼吸与危重症医学科瞿介明教授、上海交通大学生命科学技术学院微生物代谢国家重点实验室欧竑宇教授为论文的共同通讯作者,上海交通大学生命科学技术学院张剑峰博士、瑞金医院呼吸与危重症医学科徐燕萍博士、上海交通大学生命科学技术学院王萌博士为论文的共同第一作者。

doi:10.1186/s13073-023-01260-w.
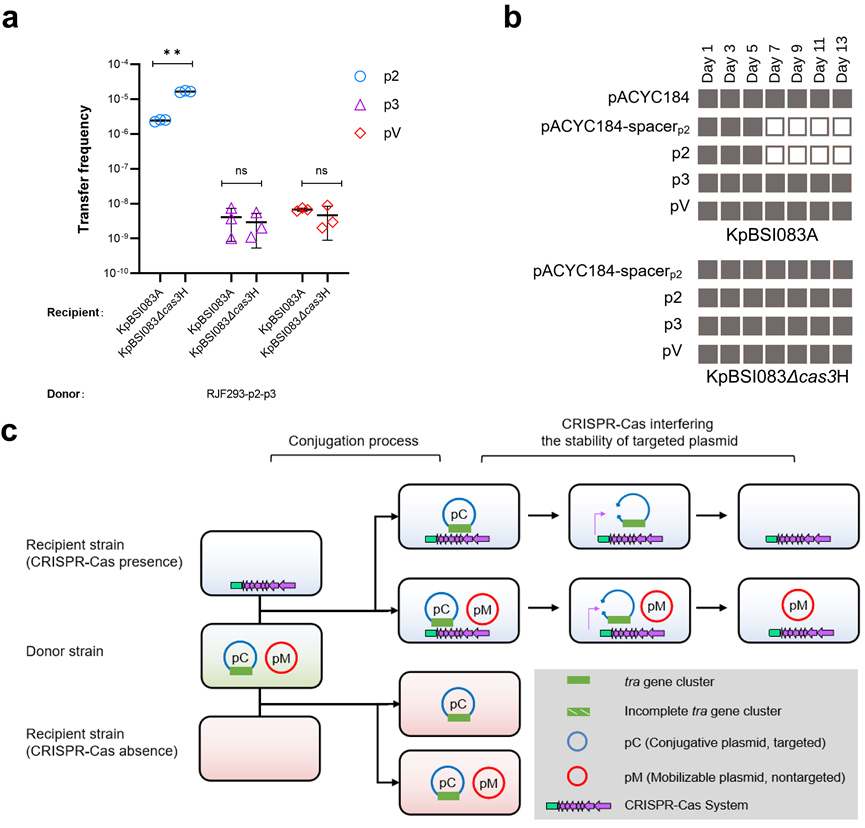
研究内容示意图
质粒是细菌致病菌中获得性抗生素耐药基因和毒力基因的主要载体,在基因水平转移中发挥着重要作用。质粒主要通过接合的方式在细菌中播散,根据其转移能力和接合机制,质粒可分为三类:接合型质粒、可诱动转移型质粒和不可转移型质粒。接合型质粒具有自主转移能力,并包含四个必需的接合元件,包括完整的四型分泌系统(T4SS)基因簇、起始转移位点(oriT)、四型伴侣蛋白(T4CP)基因和松弛酶基因。缺失部分接合元件的可诱动转移型质粒需依赖接合型质粒编码的接合元件进行非自主转移。
接合型质粒和可诱动转移型质粒约占所有质粒的50%。接合型质粒能够主动转移,且相对转移频率高,被认为是基因水平转移过程中最为重要的质粒。可诱动转移型质粒通常被认为需至少编码oriT和松弛酶。仅具有oriT的可诱动转移型质粒需依赖编码可识别和切割该oriT的同源松弛酶的特定接合型质粒的协助实现转移,这使得可诱动转移型质粒的转移条件尤为苛刻。既往有研究报道细菌对抗外源DNA的防御系统可能会影响质粒的转移,比如CRISPR-Cas系统限制了假单胞菌和克雷伯菌中接合型质粒的播散。接合型质粒的接合元件常常为CRISPR-Cas系统的靶点,使得这些质粒的宿主范围较窄。考虑到可诱动转移型质粒的接合元件有限,这些质粒可能较少受到CRISPR-Cas系统的攻击而拥有更宽的宿主范围,这些质粒对于抗生素耐药基因和毒力基因的水平转移的贡献可能被显著低估。
肺炎克雷伯菌是一种兼具多重耐药和高毒力的致病菌,在临床标本中的分离率逐渐增高。近年来,不断增长的碳青霉烯类耐药兼高毒力肺炎克雷伯菌的出现引起了医疗界的广泛重视,一直以来毒力质粒被认为是不可转移型质粒,其转移机制不明。在前期的研究工作中(Genome Medicine, 2021),研究者揭示了肺炎克雷伯菌的高毒力质粒可以借助接合型质粒发生多种形式的诱动转移,并通过构建模拟质粒、基因敲除等技术证明了毒力质粒是诱动转移型质粒,明确了毒力质粒的oriT序列。然而,肺炎克雷伯菌可诱动转移型质粒的具体转移机制及其宿主范围尚未完全阐明。
研究者通过分子生物学实验和生信分析来研究肺炎克雷伯菌临床分离株中的可诱动转移型质粒。实验中研究者观察到源自临床菌株的不编码松弛酶的可诱动转移型多重耐药质粒pKPHS3和毒力质粒pRJF293,能够在编码碳青霉烯耐药基因的接合型质粒pKPHS2的作用下,在不同的克雷伯菌菌株和大肠杆菌菌株中进行共转移。同时,研究者也观察到没有原间隔序列(CRISPR-Cas系统的靶点)的可诱动转移型质粒在受体菌中可以免受CRISPR-Cas的干扰。对三千多个质粒的生信分析显示,相比于接合型质粒,可诱动转移型质粒具有较少的原间隔序列和更广泛的宿主范围。此外,可诱动转移型质粒和接合型质粒共同促进了肺炎克雷伯菌中抗生素耐药基因和获得性毒力基因的富集。
Abstract
Background: Klebsiella pneumoniae is a notorious clinical pathogen and frequently carries various plasmids, which are the main carriers of antimicrobial resistance and virulence genes. In comparison to self-transmissible conjugative plasmids, mobilizable plasmids have received much less attention due to their defects in conjugative elements. However, the contribution of mobilizable plasmids to the horizontal transfer of antimicrobial resistance genes and virulence genes of K. pneumoniae remains unclear. In this study, the transfer, stability, and cargo genes of the mobilizable plasmids of K. pneumoniae were examined via genetic experiments and genomic analysis.
Methods: Carbapenem-resistant (CR) plasmid pHSKP2 and multidrug-resistant (MDR) plasmid pHSKP3 of K. pneumoniae HS11286, virulence plasmid pRJF293 of K. pneumoniae RJF293 were employed in conjugation assays to assess the transfer ability of mobilizable plasmids. Mimic mobilizable plasmids and genetically modified plasmids were constructed to confirm the cotransfer models. The plasmid morphology was evaluated through XbaI and S1 nuclease pulsed-field gel electrophoresis and/or complete genome sequencing. Mobilizable plasmid stability in transconjugants was analyzed via serial passage culture. In addition, in silico genome analysis of 3923 plasmids of 1194 completely sequenced K. pneumoniae was performed to investigate the distribution of the conjugative elements, the cargo genes, and the targets of the CRISPR-Cas system. The mobilizable MDR plasmid and virulence plasmid of K. pneumoniae were investigated, which carry oriT but lack other conjugative elements.
Results: Our results showed that mobilizable MDR and virulence plasmids carrying oriT but lacking the relaxase gene were able to cotransfer with a helper conjugative CR plasmid across various Klebsiella and Escherichia coli strains. The transfer and stability of mobilizable plasmids rather than conjugative plasmids were not interfered with by the CRISPR–Cas system of recipient strains. According to the in silico analysis, the mobilizable plasmids carry about twenty percent of acquired antimicrobial resistance genes and more than seventy-five percent of virulence genes in K. pneumoniae.
Conclusions: Our work observed that a mobilizable MDR or virulence plasmid that carries oriT but lacks the relaxase genes transferred with the helper CR conjugative plasmid and mobilizable plasmids escaped from CRISPR–Cas defence and remained stable in recipients. These results highlight the threats of mobilizable plasmids as vital vehicles in the dissemination of antibiotic resistance and virulence genes in K. pneumoniae.
中文摘要
背景:肺炎克雷伯菌(Klebsiella pneumoniae)是一种臭名昭著的临床病原体,常携带各种质粒,这些质粒是抗生素耐药基因和毒力基因的主要载体。与可自主转移的接合型质粒相比,可诱动转移型质粒由于其接合元件的不完整而受到较少关注。然而,可诱动转移型质粒对肺炎克雷伯菌的抗生素耐药基因和毒力基因的水平转移的贡献尚不清楚。该研究通过遗传实验和基因组分析,对肺炎克雷伯菌的可诱动转移型质粒的转移、稳定性和荷载基因进行了检测。
方法:使用肺炎克雷伯菌HS11286的碳青霉烯耐药(CR)质粒pHSKP2,多重耐药(MDR)质粒pHSKP3,以及肺炎克雷伯菌RJF293的毒力质粒pRJF293进行接合实验,评估可诱动转移型质粒的转移能力。构建模拟可诱动转移型质粒和基因修饰质粒以确认共转移模型。通过XbaI和S1核酸酶脉冲场凝胶电泳和/或完整基因组测序评估质粒形态。通过连续传代培养来分析可诱动转移型质粒在接合子中的稳定性。此外,对1194个完整测序的肺炎克雷伯菌基因组中的3923个质粒进行了基因组分析,以研究接合元件、荷载基因和CRISPR-Cas系统的靶点的分布。对携带oriT但缺乏其他接合元件的肺炎克雷伯菌可诱动转移型多重耐药质粒和毒力质粒也进行了研究。
结果:研究者的结果表明,携带oriT但缺乏松弛酶基因的可诱动转移型多重耐药质粒和毒力质粒均能够与碳青霉烯耐药的接合型辅助质粒在各种克雷伯菌和大肠杆菌菌株之间共转移。不同于接合型质粒,可诱动转移型质粒的转移和稳定性不受到受体菌中CRISPR-Cas系统的干扰。根据生信分析,肺炎克雷伯菌中的可诱动转移型质粒携带约20%的获得性抗生素耐药基因和超过75%的毒力基因。
结论:研究者的研究观察到,一个携带oriT但缺乏松弛酶基因的可诱动转移型多重耐药质粒或毒力质粒能够与碳青霉烯耐药的接合型辅助质粒共转移,并且可诱动转移型质粒能够逃避受体菌中CRISPR-Cas的攻击并在受体菌中保持稳定。这些结果凸显了可诱动转移型质粒在肺炎克雷伯菌中传播抗生素耐药基因和毒力基因的威胁。
作者介绍

瞿介明
主任医师,教授,博士生导师,上海交通大学医学院附属瑞金医院党委书记,上海交通大学医学院附属瑞金医院呼吸病研究所所长,上海市呼吸传染病应急防控与诊治重点实验室主任,中华医学会呼吸病学会主任委员;研究方向:肺部感染。

徐燕萍
医师,临床博士后,导师为瞿介明教授,上海交通大学医学院附属瑞金医院呼吸与危重症医学科;研究方向:耐药细菌的水平转移。
本文内容来源自订阅号“瑞金医院图书馆”,原链接:瑞金新知速递 第757期|瞿介明教授课题组解释肺炎克雷伯菌诱动转移型质粒促进耐药基因和毒力基因的播散
* 文章仅供医疗卫生相关从业者阅读参考
本文完
责编:Jerry